本科毕设文献翻译部分。
原作者:Chung-Chi Huang and Zhiyong Lu, National Center for Biotechnology Information (NCBI), National Library of Medicine (NLM), National Institutes of Health (NIH)
概述
竞争是一个提高 state of the art 的有效途径。随着 蛋白质结构预测竞赛(Critical Assessment of protein Structure Prediction, CASP) 在生物信息学研究中取得的成功,文本挖掘研究界组织了一系列挑战性赛事,以评估和推进生物医学相关自然语言处理(NLP)研究。在本文中,我们回顾了 2002 年至 2014 年不同的社区挑战评估及其各自的任务。此外,我们还分别通过 NLP 研究和生物医学应用中的目标问题来研究这些挑战任务。我们将简述要组织一场生物医学 NLP(BioNLP)竞赛的一般工作流程,以及涉及的利益相关者(任务组织者、任务数据生产者、任务参与者和最终用户)。最后,将不同的生物 NLP 竞赛作为一个整体,我们总结了他们的影响和贡献,然后讨论了它们的局限性和困难。我们总结了未来生物 NLP 挑战评估的趋势。
关键词
生物医学自然语言处理(BioNLP);BioNLP 竞赛;BioNLP 共享任务(shared tasks);关键性评估(critical assessment);文本挖掘
简介
大多数生物医学的最新成果都在学术刊物上发表,因此在文献中查找相关信息对于任何生命科学研究人员都是必不可少的1-4。然而,近年来生物医学文献数量迅速增长,每天(文章发表于2015年5月)在生物医学期刊上发表超过3000篇文章。这使得文献检索和信息获取成为一项艰巨的任务1 5 6。临床领域的医疗保健专业人员在处理日益增多的电子医疗/健康记录时8,也面临着类似的信息爆炸和过载问题7。
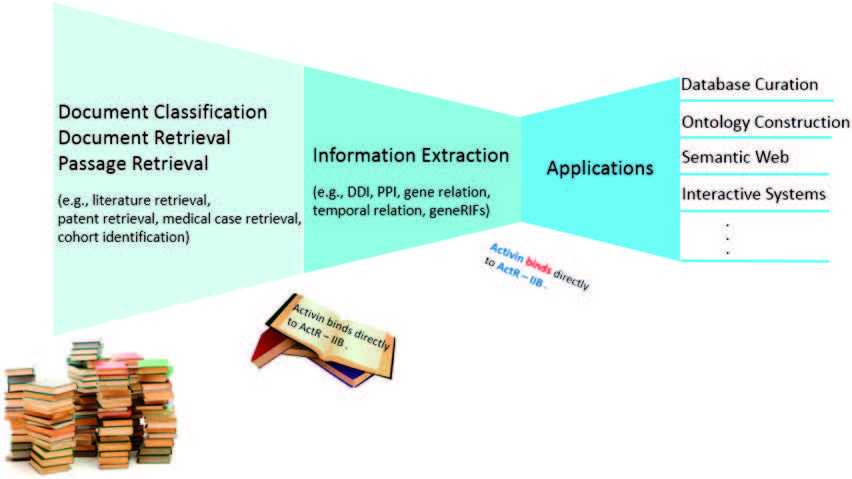
图一 数据通道技术基于 NLP 技术和通道化(channeled)数据的应用。NLP 技术(如文档收集和信息提取)帮助生命科学领域的科学家从繁重的手工选则感兴趣的文档工作中解放出来。下面举一些聚焦 NLP 的 BioNLP 竞赛的例子。例如,信息提取相关的 BioNLP 主题包括但不仅限于,寻找药物-药物相互作用、蛋白-蛋白相互作用、基因关联、临床时序关联和基因功能标注等等。通道化 / 从文本中挖掘的数据,在另一方面,可以进一步地在数据库完善、构建本体、建立语义网络等交互系统等多个方面得到落地应用。
自然语言处理(NLP)通过从文本中提取关键信息并将其转换为结构化知识以供人理解,从而可以极大地促进研究效率。由于学术出版物和临床报告主要以文本形式编写,就使其在生物医学研究中变得越来越重要9。自20世纪90年代末以来,NLP 与生物医学界的跨学科合作越来越普遍,形成了一个新的研究领域,称为生物医学自然语言处理(BioNLP)或文本挖掘,目的是为各种生物医学应用开发 NLP 方法。如 图一 所示,文本挖掘开发人员首先使用如“文档分类(document classification)”和“文档/文段收集(document/passage retrieval)”这样的信息收集(information retrieval, IE)技术来确定一部分相关文档10-16。这一步的本文选择工作,一下子将搜索范围从整个文档集缩小到了主题相关文档(相关的 BioNLP 话题包括生物医学文献检索、化学专利检索、医疗案例检索和队列识别 (cohort recognition))。这个筛选过程通常被称为文献分拣(triaging)10 17。然后,BioNLP 开发人员结合信息提取(IE)技术(例如 事件(event)提取 或 实体关系(entity-relation) 提取)来识别可能代表目标信息焦点的文本段。这个信息焦点可能是 实体-实体相互作用 (如药物-药物相互作用 (DDI) 18 和蛋白质-蛋白质相互作用 (PPI) 19)、 实体-实体关系(如蛋白质-残基关联20、基因关系21或临床记录中的时间关联22)、特定生物实体的功能或关联的参考声明或实验方法(例如基因功能标注(references into gene function, geneRIFs)23)、 生物过程 (例如磷酸化)以及 参与功能实现的生物实体 (例如基因事件 (gene event) 提取24)等。
Notes: Information Extraction Pipeline and Tasks
- Information retrieval (refining article collection)
- Focus extraction
- event extraction
- entity-relation extraction
- entity-entity interaction
- DDI / PPI
- protein-residue association
- gene relation
- temporal relation in clinical record
- references into gene function
- gene event extraction
- ……
总的来说,支持 NLP 的自动数据通道增益(the automatic NLP-enabled data channeling)使生命科学领域的用户能够接触到感兴趣的特定文本片段,而无需进行大量的手动搜索和查找。另外,从生物医学文献和临床记录中提取或文本挖掘的信息具有广泛的现实应用。例如,可以利用它来辅助数据库管理25-29、构建本体30、促进语义 Web 搜索31 32和辅助开发交互系统(例如计算机辅助校正(curation)工具33 34)。
为了促进这种跨学科的合作,同时评价和推进 NLP 技术在生物医学领域的应用,随着 CASP 对蛋白质结构预测的竞赛在 1994 年35 36的成功举办,近年来大量 BioNLP 共享任务(shared-tasks)开始举办。一些竞赛关注从生物医学文献中提取信息,而另一部分则更关注从医疗记录中提取信息。作为对主流生物医学文献知识发现工作的一点补充,本篇综述旨在简要介绍像37 38这样在 2002 到 2014 年间举办的 BioNLP 竞赛。
特别地,我们兼顾了 NLP 领域的研究和潜在的生物医学问题,并系统地总结和比较了这些比赛的主要任务和子任务。我们也简要聊了一下要举办 BioNLP 竞赛的步骤。最后,我们整体性的总结了这些竞赛的影响和贡献,同时概括了他们遇到的问题和局限性。
Notes: Article Structure
- BioNLP tasks & subtasks
- Steps to organize one
- Impacts & contributions
- Limitations & difficulties
请注意,竞赛参与体系不在本文的讨论范围内。我们希望各位读者能前去参看每个竞赛自己的任务概览介绍文章,学习参赛者们的实现方法、模型描述和模型优劣。
任务综述
图二 按时间顺序列举了 2002-2014 年间的 BioNLP 竞赛。各个竞赛用白色粗体标出,它们关注的问题用黑色斜体标注,还带了一些简要的关于 表一 中竞赛内容的注解。图二 中我们把 BioNLP 竞赛的评价按照文本类型进行了分组:关注出版文献的生物类任务和关注医疗记录的临床任务。这些竞赛任务最早在 2002 年由 ACM KDD CUP 开出先河,这些年又有 TREC Genomics 等竞赛相继举办。
Notes: BioNLP Text Genre
- Biological tasks focus on scholarly publications
- Clinical tasks on clinical records
KDD Cup, TREC Genomics/Chemical 和 CoNLL
早年间的竞赛,如 KDD Cup 和 TREC Genomics,主要关注文本收集23和文本分类17任务。比如 KDD Cup 2002 年39的 the fly genetics task 需要参赛者判断是否一篇文章符合苍蝇基因表达的各项准则。
TREC ad hoc 2003 年的竞赛任务23 需要参与者收集基因功能相关的文献(例如选取描述基因功能的文献),并且接下来两年的任务是收集基因和其他生物实体相关的文献。TREC 也尝试了23基因功能(GeneRIFs)相关的文段 / 表述提取41 42。在随后的的 2006 年43和 2007 年44,
TREC Genomics 进一步将搜索主题制定为自然语言问题(2007年基于生物实体的问题),通过NLP解决生物学家在问答(QA)范例中的相关任务。 继续以前在文件检索方面的努力,TREC 在 2009 年至 2011 年期间组织了一条化学试剂赛道45-47,以满足化学专利中文件筛选的需求。 对于文本分类,CoNLL 将其 2010 年的共同任务专门用于识别生物医学文献中的不确定句子和定位句内套头线索(hedging cues)48,因为生物医学出版物中常见的否定和推测可能对文本挖掘结果产生直接影响。
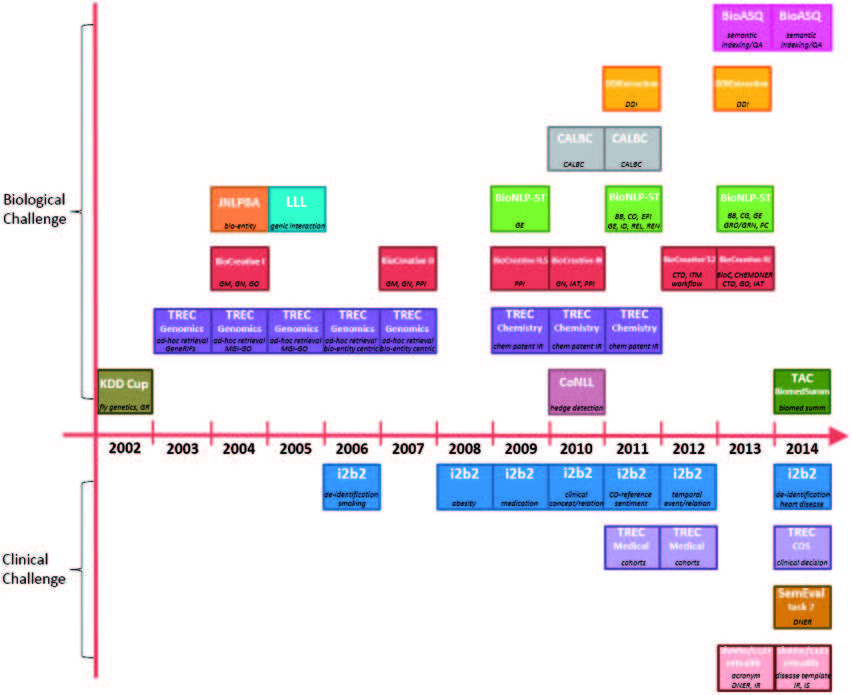
图二 BioNLP 竞赛按时间顺序排列。竞赛以粗体白色字体显示,而其在表一中的关注任务/赛道以斜体黑色字体显示。
BioCreative, JNLPBA 和 CALBC
2004 年,BioCreative 和 JNLPBA 开始把重心放到自动检测自由文本(free text)中生物实体上。具体而言,BioCreative I50 中的 Gene Mention(GM)任务49旨在检测基因名称,而 JNLPBA 51中的生物实体任务涉及多种实体类型,如 DNA、RNA 和细胞类型。生物命名实体识别(NER)是必不可少的,因为它是许多高级NLP任务的构建模块,如蛋白质 - 蛋白质相互作用或基因调控(GR)提取51。在GM任务之后,引入BioCreative I 52,BioCreative II 53和BioCreative III 54中的基因标准化(GN)任务,其中自动定位的基因名称进一步映射到某些标准词典/数据库中的唯一标识符(例如 EntrezGene)。除了基因或蛋白质,最近的 BioCreative 任务55-57也研究了其他关键生物实体(如化学品和疾病)的自动检测。 CALBC 是另一个面向 NER 的挑战,目标是生成一个带有注释生物实体的大型共享语料库58 59。
KDD Cup, LLL, BioCreative, DDIExtraction 和 BioASQ
同时,IE 技术已经在各种生物相关主题中进行了测试,例如 2002 年 KDD 杯中的 GR 预测赛道和 LLL 2005 中的基因相互作用提取任务21。在 BioCreative 中,引入了两个主要的 IE 任务:基因本体术语的自动分配(即 BioCreative I60 和 IV61 62 中的 GO 任务)和蛋白质-蛋白质相互作用的提取(即 PPI 任务) BioCreative II19,II.563 和 III12)。 2011 年,DDI 任务首次引入 DDIExtraction18,然后在 2013 年重复64。面对与BioCreative中的 GO 任务类似的挑战,由于 MeSH 词典的大小和文本中 MeSH 概念的丰富表达(可能涉及词义消歧),因此将 MeSH 术语自动分配给生物医学文章本身65更具挑战性。因此,它成为最近 BioASQ 挑战中的两项任务之一66 67,而另一项任务则侧重于从现实生物医学研究中获得对问题的精确和可理解的答案。
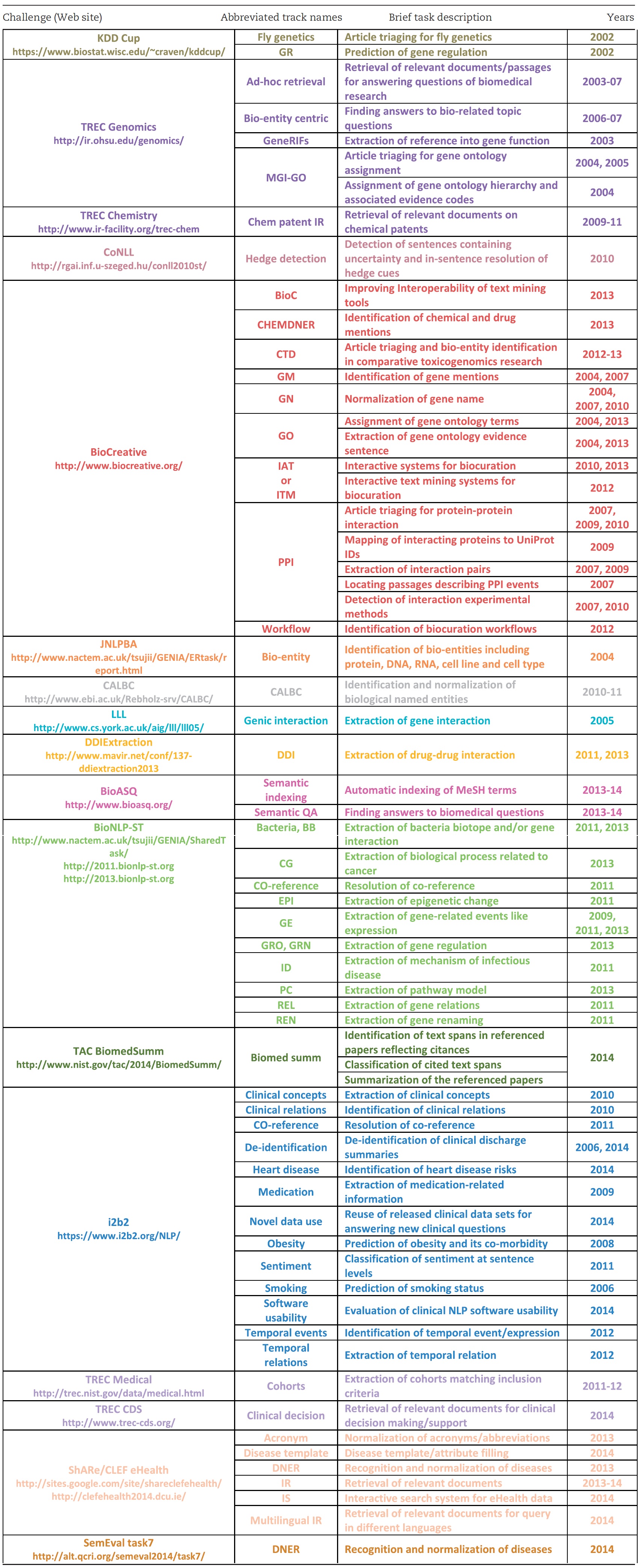
表一 根据在本文中的出现顺序排列的各项竞赛,竞赛的任务按缩写名称的字母顺序排列。 文本采用与图二一致的颜色编码。该表的彩色版本可在 BIB 网站上获得:http://bib.oxfordjournals.org。
BioNLP-ST
与 BioCreative 任务和其他面向 IE 的赛事相比,BioNLP-ST 在如何与参与实体一起表示生物事件/过程具有独特的语义,并致力于事件/关系提取。例如,2009 年24,2011 68和 2013 69的 BioNLP-ST GENIA(GE)任务要求任务参与者提取基因相关事件,如调节、表达和转录,并将它们相应关联。活动参与者,本地化或网站。 BioNLP-ST 2011 细菌任务70和 2013 Bacteria Biotope 任务71旨在检测细菌的栖息地,而 BioNLP-ST 2011 传染病任务72和 2013 年癌症遗传学(CG)任务73分别针对生物分子传染病和癌症遗传学的机制。
BioNLP-ST 还涵盖了如代谢途径管理(即BioNLP-ST 2013 74中的Pathway Curation任务)这样高层次和共引用冲突解决(即BioNLP-ST 2011 75中的共引用(CO参考)和名称别名(即BioNLP-ST 2011中的REN任务76)这样基础的任务。这些问题虽基本,但恰恰是共同引用和别名问题明显地对事件提取系统的性能设定了天花板。基因调控(例如 2013 年的 GRO 和 GRN 任务77)和基因互作(例如 2011 年的细菌任务70)也得到了解决。
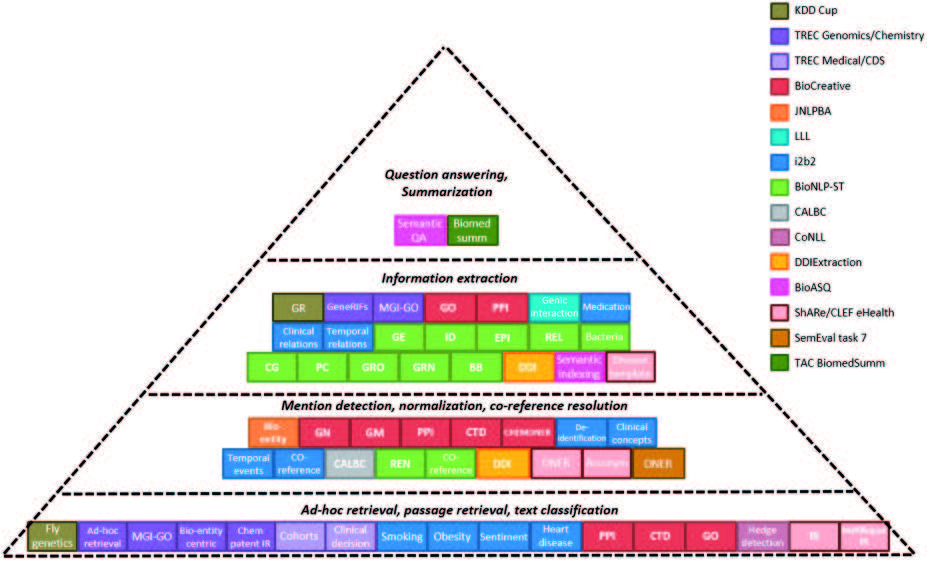
图三 按 NLP 领域分类的竞赛子任务和赛道。该图的彩色版本可在 BIB 网站上获得:http://bib.oxfordjournals.org。
TAC BiomedSumm
2014 年,TAC BiomedSumm 赛道要求参与者利用引用特定论文(“citances”)的引用句子集进行综述(summarization),这是 BioNLP 研究中的一个重要问题42 78。 具体而言,该赛道包括识别反映参考文章的英文内容文本段,将这些文本段分类为各个文章论点,然后基于其社区对其引用的讨论生成所引用论文的摘要。
i2b2, TREC Medical/CDS
第一个临床导向的挑战任务由 Informatics for Integrating Biology and the Bedside(i2b2)在 2006 年发起。早期的重点是“去标识”79,这是一项与 NER 相似的任务,因为医疗中敏感的私人健康信息 / 临床记录需要在分发前删除。同年,i2b2 举办了在文献(如临床记录)水平确定吸烟状况80的文本分类任务,预测 2008 年文献(如临床记录)中肥胖及其并发症的水平81、在2011年的句子层面(来自自杀笔记)确定情绪水平82,以及预测 2014 年文献(如临床记录)层面的心脏病风险。i2b2 也对提及检测和概念识别感兴趣,但对生物实体不感兴趣。相反,他们在 2009 年83和 2010 84 以及临床叙述中解决了临床概念,例如医学问题,测试,治疗,药物和剂量,以及 2012 年的时间相关表达22。然后,在 2010 年,通过断言信息(例如,是否存在医学问题)对已识别的实体进行分析84,或者在 2012 年后与时序关系(例如手术前的剂量)相关联22。
另一方面,TREC近年来将重点从生物医学文献转向临床记录。 TREC 医疗跟踪于 2011 年85和 2012 年86引入,旨在确定符合特定“纳入标准”(例如性别,年龄组,治疗和疾病)的队列,用于临床研究,临床试验或流行病学研究。 2014 年,TREC CDS跟踪研究了用于临床决策支持的医疗案例检索的 NLP 技术。
ShARe/CLEF eHealth 和 SemEval
除了 i2b2 和 TREC Medical / CDS 之外,2013 年还开展了一项名为 ShARe / CLEF eHealth 的新评估赛事87。它涉及三个单独的任务(a)临床记录和标准化中疾病名称的传统 NER,(b)将临床文件中的首字母缩略词和缩写映射到 UMLS CUI,以及(c)检索相关文件以解决患者在阅读出院摘要时可能遇到的问题。 2014年,SemEval 88的任务7重复了疾病 NER 和 ShARe / CLEF eHealth 2013 的常规任务,而 ShARe / CLEF eHealth 2014 推出了一系列不同的任务89 :(a)电子卫生数据交互式搜索系统,(b)疾病模板 / 属性填充90和(c)临时医疗记录检索91,其中任务(c)是首次尝试处理多种语言。
在图三中,我们通过 NLP 研究中的目标问题对表一中的挑战赛道进行分类:从 IR(临时检索,段落检索和文本分类)到 NER(提及检测,归一化和共引用),到 IE 以及质量保证和总结。例如,TREC Medical 的队列跟踪属于类别文本分类,而 BioCreative 的 CHEMDNER 跟踪92提及检测(化学,药物和疾病检测准确),其 GM 和 GN 跟踪也是如此。一般而言,靠近金字塔顶部的 NLP 任务更加困难。从图三中我们可以看出,在共享任务中,TREC 非常关注 IR,而其他(例如 BioCreative,LLL,BioNLP-ST 和 DDI)则关注 NER 和 IE。
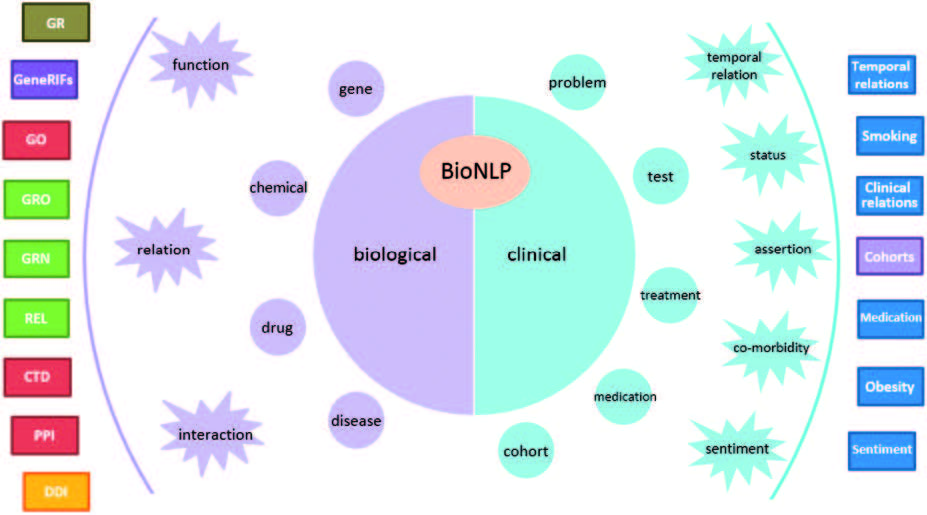
图四 BioNLP 针对的不同生物和临床问题相关竞赛。 挑战子任务以与图二中相同的颜色编码(例如,BioNLP-ST 任务是绿色标记的)。 该图的彩色版本可在BIB网站上获得:http://bib.oxfordjournals.org。
从不同的角度来看,图四根据生物学和医学中的目标问题分类了 BioNLP 的各项竞赛。
可以看出,这两个领域有自己感兴趣的实体和关系,分别用图四中的圆圈和爆炸表示。在生物学方面,关键的生物实体包括基因 / 蛋白质、化学物质、药物和疾病93-96;而在临床方面,医疗问题、测试、治疗、药物相关信息和队列最受关注。至于重要关系,生物学任务主要针对生物实体的功能(例如 BioCreative 的 GO 任务中的基因功能)、关系事件(例如 BioNLP-ST 的 GE 中的基因相关事件)和相互作用(例如药物-药物作用)。在 DDI 提取的 DDI 任务中的相互作用和 BioCreative 的 PPI 任务中的蛋白质-蛋白质相互作用。另一方面,临床任务涉及临床实体之间完全不同的关系集,例如时间关系、状态、断言/风险、合并症和情绪分析。图四尽可能将各个任务(在框中)与其关联实体和关系对齐。例如,i2b2 肥胖任务81与共病的关系一致,因为它旨在预测肥胖医学问题的共病,而 i2b2 时间任务[22]与时间关系一致,其目标是确定医学概念(例如治疗和问题)之间的时间关系。
如何举办一个竞赛
这部分我不是很感兴趣所以全部跳过了,感兴趣的朋友移步 原文。
正式竞赛评估的局限性
首先,由于开放式评估的性质(任务必须明确定义并具有“适当的”难度级别),挑战任务总是被简化或从现实问题中抽象出来。例如,由于访问受限制和处理全文的困难,许多 BioNLP 挑战任务共同的简化步骤是使用摘要,尽管个别研究人员,数据索引者和策展人经常阅读全文 61 65。其他例子包括在 QA 或 IR 任务中使用适度数量的人为和结构良好的有限类型问题,而在现实中,信息搜索者通常会提出复杂且开放式的自然语言问题,这些问题往往是形式错误和不合语法的3 。
其次,算法开发可能没有足够的参与度和创新。由于任务本身(太难或没有吸引力)或同时由于其他竞争任务而导致参与不足。从图二中可以看出,近年来总是有比单个群体可以解决的更多并发任务。结果,一些任务的参与率很低(少于五个)。此外,我们注意到,当一项任务被分解为多个子任务时,更少的团队可以选择端到端地完成任务。例如,在 BioNLP-ST 2009 24中,有 24 个团队参与了 GE subask-1,尽管只有两个团队完成了所有三个子任务。在技术进步方面,挑战任务旨在通过开发新的和不同的方法来刺激研究界的进步。然而,当发现现有方法有效且具有竞争力时,我们经常发现团队方法缺乏多样性。例如,2006 年 i2b2 挑战中吸烟状态检测任务的前 12 个系统中,9 个使用支持向量机,其微量平均 F-measures 没有统计学上的显着差异80。
最后,挑战任务与实际使用之间存在差距也有着许多的原因。首先,对于某些任务,即使最好的自动化结果也明显低于实际环境中所需的结果。此外,如前所述,挑战任务总是实际问题的抽象,因此即使是高性能系统也会受到现实世界应用中的许多其他因素(例如,不同的文本输入,系统可扩展性或互操作性)的困扰。例如,之前的 BioCreative Gene Normalization 挑战表明,在全文54而不是摘要53上测试时,任务性能显着下降。最后,许多参与此类挑战的参与者的传统是实现竞争性绩效并在声誉良好的期刊上发布结果。直到最近,团队几乎没有动力去适应并将他们的方法推向现实世界。
未来趋势
我们觉得鉴于现阶段的成功,在未来竞赛性的评估会继续在 BioNLP 领域中扮演非常关键的作用。虽然一些基本任务(例如命名实体识别和信息提取等)仍将继续,同时我们也非常期待能在不远的将来看到,以不同生物、临床中用户的需求为重点的项目得以举办。我们也希望,成功有效的方法和技术能持续不断地转化到实际应用中117 118。向着这个目标,除系统准确性以外,系统扩展性和互用性(interoperability)也需要在未来的竞赛评估中得到进一步的重视119 120。另一个趋势可能是,在给定资源有限的条件下,与其各自分散举办不同的竞赛,不如搭建一个更加协同、更加高效、更加的经济的竞赛框架,其可以同时惠及竞赛参与者和组织者。最后,真实的竞赛评估已经在一定程度上展现出其实用性,但是协同性的竞赛可能是共同解决单个队伍无法解决的现实问题的最终途径。
本文提要
- 我们回顾了 2002 年到 2014 年的生物学和临床领域的 BioNLP 竞赛。
- 我们从 NLP 和实体关联的角度总结了各项竞赛的子任务。
- 我们描述了学术社区竞赛评估举办的几个步骤。
- 我们从各个角度讨论了各项 BioNLP 竞赛的影响、贡献和各自面临的问题和局限性。
参考文献
(此处仅链接前十篇,其余文章请前往 原文 查阅)
1. Lu Z. PubMed and beyond: a survey of web tools for searching biomedical literature[J]. Database, 2011, 2011. ↩
2. Khare R, Leaman R, Lu Z. Accessing biomedical literature in the current information landscape[M]//Biomedical Literature Mining. Humana Press, New York, NY, 2014: 11-31. ↩
3. Islamaj Dogan R, Murray G C, Névéol A, et al. Understanding PubMed® user search behavior through log analysis[J]. Database, 2009, 2009. ↩
4. Jensen L J, Saric J, Bork P. Literature mining for the biologist: from information retrieval to biological discovery[J]. Nature reviews genetics, 2006, 7(2): 119. ↩
5. Shatkay H, Feldman R. Mining the biomedical literature in the genomic era: an overview[J]. Journal of computational biology, 2003, 10(6): 821-855. ↩
6. Hunter L, Cohen K B. Biomedical language processing: what’s beyond PubMed?[J]. Molecular cell, 2006, 21(5): 589-594.00114-6) ↩
7. Berner E S, Moss J. Informatics challenges for the impending patient information explosion[J]. Journal of the American Medical Informatics Association, 2005, 12(6): 614-617. ↩
8. Linder J A, Ma J, Bates D W, et al. Electronic health record use and the quality of ambulatory care in the United States[J]. Archives of internal medicine, 2007, 167(13): 1400-1405. ↩
9. Cohen K B, Hunter L. Natural language processing and systems biology[M]//Artificial intelligence methods and tools for systems biology. Springer, Dordrecht, 2004: 147-173. ↩
10. Hersh W, Bhuptiraju RT, Ross L, et al. TREC 2004 genomics track overview. In: Proceedings of the 13th Text Retrieval Conference, 2004 ↩
(此处仅链接前十篇,其余文章请前往 原文 查阅)